250x250
Recent Posts
Recent Comments
Archives
- Today
- Total

KimDove
안녕하세요, 딥러닝 엔지니어 김둘기 입니다.
비둘기 둥지
[생물정보학 / python] 3-1. 지금까지 공부한 내용 연습 본문
728x90
1. 캐글 데이터 셋으로 분석해보기
from Bio.Align.Applications import MuscleCommandline
from Bio.SeqUtils import MeltingTemp as mt
from Bio.SeqRecord import SeqRecord
from Bio.SeqUtils import GC
from Bio.Seq import Seq
from Bio import SeqIO
import re
## Kaggle에서 제공된 데이터 셋이 txt 파일로 되어 있어
## SeqIO가 아닌 파이썬의 파일 입출력으로 염기서열을 가져옴.
def text_parsing(spieces):
sequence_txt = open(f'../dataset/txt/{spieces}_data.txt', 'r').readlines()
pattern = re.compile('[AGCT]+')
sequences = []
seq_append = sequences.append
## text 파일 맨 첫번째 줄에 sequence class 이런 데이터가 있어 두번째 줄 부터 가져옴.
for sequence in sequence_txt[1:]:
## 두번째 줄부터 문자열 끝부분에 숫자가 붙어있어 정규표현식으로 염기서열(A, G, C, T)만 가져옴.
match = re.findall(pattern, sequence)
seq_append(match)
return sequences
sequences = [
text_parsing('human')[0][0],
text_parsing('dog')[0][0],
text_parsing('chimpanzee')[0][0]
]
longest_length = max(len(seq) for seq in sequences)
## 가장 긴 염기서열 길이만큼 맞춤.
## 가장 긴 염기서열보다 길이가 짧으면 -으로 채움.
pad_seq = [seq.ljust(longest_length, '-') for seq in sequences]
names = ['human', 'dog', 'chimpanzee']
for name, record in zip(names, pad_seq):
print(f'{name} \n염기 : {record[-15:]} \nGC 함유량 : {GC(record):.3f}')
print(f'녹는점 : {mt.Tm_Wallace(record):.3f}, 길이 : {len(record)} (bp)\n')
records = (SeqRecord(Seq(seq), id = name, name = name) for seq, name in zip(pad_seq, names))
## 출력 결과
human
염기 : CCCCCACAATCCTAG
GC 함유량 : 39.614
녹는점 : 578.000, 길이 : 207 (bp)
dog
염기 : CCCCCTCAATAA---
GC 함유량 : 34.783
녹는점 : 552.000, 길이 : 207 (bp)
chimpanzee
염기 : CCCCCACAATCCTAG
GC 함유량 : 40.097
녹는점 : 580.000, 길이 : 207 (bp)## fasta 파일로 저장
SeqIO.write(records, '../dataset/fasta/practice.fasta', format = 'fasta')
## MUSCLE로 aln파일 저장
muscle_path = '../utils/muscle'
cmd_line = MuscleCommandline(muscle_path, input = '../dataset/fasta/practice.fasta',
out = '../dataset/aln/msa.aln', clw=" ")
std_out, std_err = cmd_line()
## MUSCLE 실행한 결과
MUSCLE (3.8) multiple sequence alignment
dog ATGCCACAGCTAGATACATCCACCTGATTTATTATAATCTTTTCAATATTTCTCACCCTC human ATGCCCCAACTAAATACTACCGTATGGCCCACCATAATTACCCCCATACTCCTTACACTA chimpanzee ATGCCCCAACTAAATACCGCCGTATGACCCACCATAATTACCCCCATACTCCTGACACTA ***** ** *** **** ** ** * ***** * *** * ** ** **
dog TTCATCCTATTTCAACTAAAAATTTCAAATCACTACTACCCAGAAAACCCGATAACCAAA human TTCCTCATCACCCAACTAAAAATATTAAACACAAACTACCACCTACCTCCCTCACCAAAG chimpanzee TTTCTCGTCACCCAACTAAAAATATTAAATTCAAATTACCATCTACCCCCCTCACCAAAA ** ** * *********** * *** * **** * ** * * **
dog TCTGCTAAAATTGCTGGTCAACATAATCCTTGAGAAAACAAATGAACGAAAATCTATTCG human CCCATAAAAATAAAAAATTATAACAAACCCTGAGAACCAAAATGAACGAAAATCTGTTCG chimpanzee CCCATAAAAATAAAAAACTACAATAAACCCTGAGAACCAAAATGAACGAAAATCTATTCG * ***** * * ** ** ****** **************** ****
dog CTTCTTTCGCTGCCCCCTCAA---TAA human CTTCATTCATTGCCCCCACAATCCTAG chimpanzee CTTCATTCGCTGCCCCCACAATCCTAG **** *** ******* *** **## WebLogo 그려보기
from Bio.motifs import Motif
from Bio import AlignIO
from Bio import motifs
sequences = AlignIO.read('../dataset/aln/msa.aln', format = 'clustal')
proteins, alphabets = [], set()
protein_append = proteins.append
for sequence in sequences:
alphabets = alphabets.union(set(sequence.seq))
protein_append(sequence.seq)
alphabets = ''.join(alphabets)
m = motifs.create(proteins, alphabet = alphabets)
Motif.weblogo(m, '../Weblogo/human_chimpanzee_and_dog.png')
## 주피터 내에서 생성한 Weblogo 파일 보이기
from IPython.display import Image
Image('../Weblogo/human_chimpanzee_and_dog.png')
## 출력 결과
Weblogo 파일이 제대로 저장되지 않았다 ㅠㅠㅠㅠㅠㅠㅠㅠㅠ## 계통수 표현해 보기
from Bio import Phylo
tree = Phylo.read('../dataset/newick/h_c_d.newick', 'newick')
print(tree)
Phylo.draw(tree)
## 출력 결과
Tree(rooted=False, weight=1.0)
Clade(branch_length=0.265821)
Clade(branch_length=0.265821)
Clade(branch_length=0.0462122, name='human')
Clade(branch_length=0.0462122, name='chimpanzee')
Clade(branch_length=0.265821, name='dog')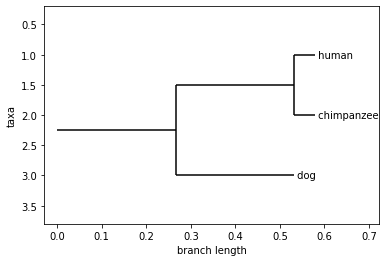
- 3개의 종으로만 분석해 본 결과, 공통 조상으로부터 (개), (인간, 침팬지)로 나뉘었고,
그 후 인간, 침팬지로 분화 되었음을 보임
99. 자료 출처
99-1. 도서
99-2.웹 사이트
- 길이가 다른 sequence를 multiple sequence alignment | stackoverflow [페이지 링크]
- 인코덤 | Biopython/PhyIo [페이지 링크]
- 유럽 생물정보학 연구소 | phylogeny input example [페이지 링크]
99-3. 데이터셋
- 인간 DNA 데이터 | Kaggle(by Neel Vasani) human-dna-data [데이터 셋 링크]
- 강아지, 침팬지 데이터 | Kaggle(by Neel Vasani) chimpanzee and dog dna [데이터 셋 링크]
전체코드
GitHub - EvoDmiK/TIL: Today I Learn
Today I Learn. Contribute to EvoDmiK/TIL development by creating an account on GitHub.
github.com
내용 추가 이력
부탁 말씀
개인적으로 공부하는 과정에서 오류가 있을 수 있으니, 오류가 있는 부분은 댓글로 정정 부탁드립니다.
728x90
'생물 정보학 > python' 카테고리의 다른 글
| [생물정보학 / python] 3.biopython - Multiple Sequence Alignment (0) | 2022.04.14 |
|---|---|
| [생물정보학 / python] 2.biopython - SeqRecord 객체, 다양한 데이터 포맷 조작 (0) | 2022.04.10 |
| [생물정보학 / python] 1.biopython - Sequence 객체 조작 (2) | 2022.03.30 |
Comments


