인공지능 공부/Pytorch
[인공지능 기초 / pytorch] 5. 그래프와 pytorch_geometric
KimDove
2022. 11. 24. 18:22
728x90
1. 그래프 (Graph)
1-1. 그래프 (Graph)란
- 그래프(Graph)는 노드와 간선으로 이루어진 구조
- 아래 이미지는 G = {{1, 2, 3}, {{1,2}, {2, 3}, {1, 3}}}으로 정의된 그래프이다.
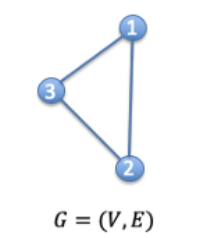
[사진 1] 간단한 구조의 그래프
- 아래 이미지는 G = {{1, 2, 3}, {{1,2}, {2, 3}, {1, 3}}}으로 정의된 그래프이다.
- 수학적으로 그래프 G는 노드 집합 V와 간선 집합 E로 구성된다.
- 간선 집합 E의 원소 간선 e는 두 개의 노드로 이루어진 순서가 있는 쌍이다.
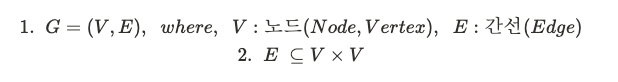
[사진 2] 그래프의 수학적 정의
- 간선 집합 E의 원소 간선 e는 두 개의 노드로 이루어진 순서가 있는 쌍이다.
- 두 간선 (a, b)와 (b, a)가 있다고 가정할 때,
- 방향성 그래프 (directed graph) | 두 간선을 같은 것으로 봄.
- 비방향성 그래프 (undirected graph) | 두 간선을 다른 것으로 봄.
❗방향성의 그래프를 시각화 할 때, 방향성은 화살표로 표시한다.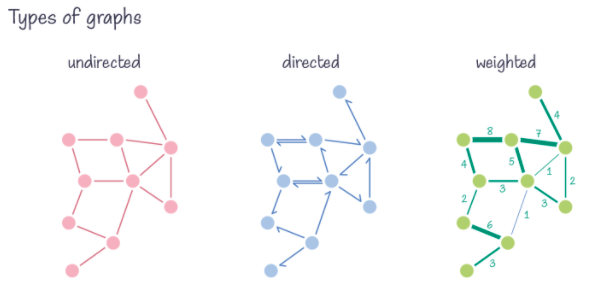
[사진 3] 그래프의 종류
- 그래프는 인접행렬로도 표현할 수 있는데, 그래프가 N개의 노드를 가지고 있다면 N x N 인접행렬이 구성된다.
- 각 노드별 feature의 갯수가 f 개인 경우에는 N x f feature 행렬이 구성된다.
⚠️ e.g.)
G = {{1, 2, 3}, {{1, 2}, {2, 3}, {1,3}}}로 정의되고, 각각의 노드가 {[1, 1], [0, 1], [1, 0]}의 feature를 가진다고 하면,
1. 그래프 G의 인접 행렬
| node | 1 | 2 | 3 |
| 1 | 0 | 1 | 1 |
| 2 | 1 | 0 | 1 |
| 3 | 1 | 1 | 0 |
| feature1 | feature2 | |
| node1 | 1 | 1 |
| node2 | 0 | 1 |
| node3 | 1 | 0 |
1-2. 그래프를 사용하는 이유
- 사람들간의 관계, 상호작용같은 추상적인 개념을 다루기에 적합하다.
- 복잡한 문제를 더 간단한 표현으로 단순화하기도 하고, 다른 관점으로도 표현하여 해결할 수 있다.
- 소셜 네트워크, 미디어의 영향, 바이러스 확산 등을 연구, 모델링 할 수 있다.
1-3. 그래프를 분석하기 어려운 이유
1) 유클리드 공간에 존재하지 않아 좌표계에서 그래프를 표현할 수 없다.
- 우리에게 익숙한 좌표계에서 표현할 수 없어 이미지나 정형데이터보다 분석이 어렵다.

2) 그래프는 고정된 형태로 존재하지 않는다.
- 동일한 인접행렬을 가진다 해도, 그래프는 무궁무진한 형태로 표현될 수 있다.
- 아래 두 그래프의 생김새는 달라도 인접행렬은 동일한 것을 볼 수 있다.

[사진 5] 그래프 모양은 다르지만, 인접행렬은 같은 그래프의 예시
| node | 1 | 2 | 3 | 4 | 5 |
| 1 | 0 | 1 | 1 | 0 | 0 |
| 2 | 1 | 0 | 1 | 0 | 0 |
| 3 | 1 | 1 | 0 | 1 | 1 |
| 4 | 0 | 0 | 1 | 0 | 1 |
| 5 | 0 | 0 | 1 | 1 | 0 |
3) 그래프는 사람이 해석할 수 있도록 시각화 하는것이 어렵다.
- 노드의 개수가 많고 조밀할수록 그래프를 보고 해석하기 힘들어진다.
1-4. 전통적인 그래프 분석법
| 알고리즘 | 예시 |
| 탐색 알고리즘 | DFS, BFS 등 |
| 최단 경로 알고리즘 | Dijkstra's Algorithm, Nearest Neighbour |
| 최소 신장 트리 알고리즘 | Prim's Algorithm |
| 클러스터링 기법 | Highly Connected Components, K-means |
- 위 알고리즘들은 적용하기 전 입력 그래프에 대한 사전지식이 필요하다는 한계가 있다.
- 여러개의 그래프가 있을때 그래프의 정보를 예측하는 것이 불가능하다.
2. Node Embedding (정점 표현)
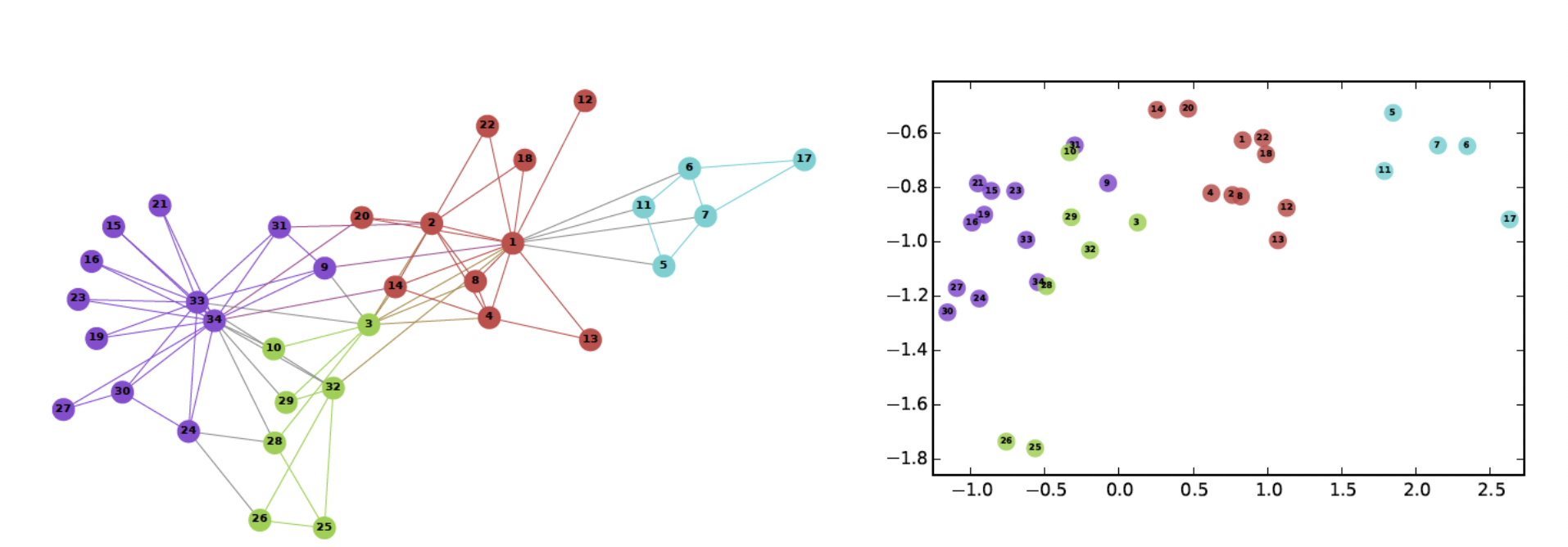
- 그래프의 node들을 벡터화 하는 것을 Node Embedding이라 한다.
- node가 표현되는 vector 공간을 embedding 공간이라한다.
- 비 유클리드 공간에서의 그래프 형태가 아닌, 유클리드 공간에서의 벡터로 처리가 가능해져
여러 툴과 프레임워크에서 사용할 수 있게된다. - 그래프에서의 정점간 유사도를 embedding 공간에서도 보존하는 것을 목표로 한다.
⚠️ e.g.)
- 두 노드를 각각 u, v라고 하고 embedding된 두 노드를 각각 𝑧𝑢, 𝑧𝑣라고 한다면,
embedding 공간에서의 유사도는 아래와 같다.
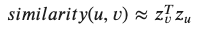
2-1. Transductive method (변환식 방법)
- node 각각에 대한 embedding vector를 얻는 방법
- Deepwalk, Node2vec이 대표적인 기법이다.
- 한계점
1) 이후 추가된 node에 대해서는 embedding을 얻을 수 없다.
2) 모든 node에 대한 embedding을 모두 계산하여 저장해두어야 한다.
3) node가 속성(Attribute) 정보를 가진 경우 이를 활용할 수 없다.
2-2. Inductive method (귀납식 방법)
- 변환식 빙법의 한계를 보완하기 위해 나온 방법으로,
각 node를 embedding vector로 변환시키는 encoder 자체를 얻는 방법이다. - Latent Factor Model, GNN 등이 대표적인 기법이다.
3. Pytorch Geometric (PyG)
- Graph와 PointCloud, Manifold에 대한 딥러닝을 가능하게 하는 Pytorch 라이브러리
- Graph와 관련된 신경망이 구현되어 있으며, GPU의 고속 활용이 가능하다.
- 사용하는 파이썬과 CUDA버전에 맞게 PyG들의 디펜던시들도 설치해줘야하는 귀찮음이 있다..
!nvcc -V
!python -V
import torch
torch.__version__
## 출력 결과
nvcc: NVIDIA (R) Cuda compiler driver
Copyright (c) 2005-2021 NVIDIA Corporation
Built on Sun_Feb_14_21:12:58_PST_2021
Cuda compilation tools, release 11.2, V11.2.152
Build cuda_11.2.r11.2/compiler.29618528_0
Python 3.7.15
1.12.1+cu113!pip install torch_geometric
!pip install https://data.pyg.org/whl/torch-1.12.0%2Bcu113/torch_sparse-0.6.15-cp37-cp37m-linux_x86_64.whl
!pip install https://data.pyg.org/whl/torch-1.12.0%2Bcu113/torch_scatter-2.0.9-cp37-cp37m-linux_x86_64.whl
## 출력 결과 생략3-1. 그래프 데이터 핸들링
- Pytorch Geometric에서 하나의 그래프는 torch_geometric.data.Data라는 클래스로 표현되고,
다음과 같은 속성값들을 갖는다.
⚠️ 아래의 인자값들은 옵션으로, 자신이 구성하고 싶은 속성을 다양하게 모델링 할 수 있다.
| 인자값 | 설명 |
| x | 노드의 특징 행렬 |
| edge_index | 그래프의 연결성 (인접행렬) |
| edge_attr | 엣지 특징 행렬 |
| y | target 값 |
| pos | 노드 위치 행렬 |
- Data를 통해 그래프를 정의하는 방법은 두 가지가 있다.
1) 𝑛×2 형태의 텐서로 정의하는 방법
2) (v1, v2) 순서 쌍으로 정의하는 방법
→ edge_index를 전치시켜 1)번과 같은 형태로 만든 후,
contiguous() 메소드를 통해 메모리 주소값을 초기화 해준다.
⚠️ 엄밀히 말하면 메모리 주소값을 초기화하는 것은 아니고, 내용이 길어서 따로 정리하도록 하겠습니다.
- 간단한 예시로 아래 이미지같은 그래프처럼 노드 0, 1, 2로만 구성된 그래프를 만들어보자
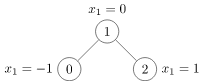
[사진 9] 간단한 그래프를 PyG로 만들어보자
## 1번 방법
## 그래프의 연결 정보
edge_idx = torch.tensor([[0, 1, 1, 2],
[1, 0, 2, 1]], dtype = torch.long)
## 노드들의 특징 행렬
x = torch.tensor([[-1], [0], [1]], dtype = torch.float)
data = Data(x = x, edge_index = edge_idx)
## 2번 방법
edge_idx = torch.tensor([[0, 1],
[1, 0],
[1, 2],
[2, 1]], dtype = torch.long)
x = torch.tensor([[-1], [0], [1]], dtype = torch.float)
data2 = Data(x = x, edge_index = edge_idx.t().contiguous())
data, data2
## 출력 결과
Data(x=[3, 1], edge_index=[2, 4]), Data(x=[3, 1], edge_index=[2, 4])- torch_geometric.data.Data에서는 그래프와 관련된 유용한 속성과 메소드도 제공한다.
| 함수명 | 설명 |
| data.keys | 해당 속성 이름 |
| data.num_nodes | 총 노드 개수 |
| data.num_edges | 총 엣지 개수 |
| data.num_node_features | 노드 특성 개수 |
| data.has_isolated_nodes() | 고립 노드 여부 확인 |
| data.has_self_loops() | 셀프 루프 포함 여부 확인 |
| data.is_directed() | 그래프의 방향성 여부 확인 |
print(f'''data keys : {data.keys}
number of node features : {data.num_node_features}
number of nodes : {data.num_nodes}
number of edges : {data.num_edges}\n''')
print(f'has isolated nodes? : {data.has_isolated_nodes()}')
print(f'has self loops? : {data.has_self_loops()}')
print(f'is graph directed? : {data.is_directed()}')
## 출력 결과
data keys : ['edge_index', 'x']
number of node features : 1
number of nodes : 3
number of edges : 4
has isolated nodes? : False
has self loops? : False
is graph directed? : False3-2. PyG에 있는 데이터 셋을 불러와보자.
- 많은 딥러닝, 머신러닝 프레임워크에서 공통 벤치마크 데이터 셋이 있는 것처럼,
PyG에도 TUDataset같은 Graph, 3d 관련 task에서 사용되는 공통 벤치마크 데이터 셋이 존재한다. - 그 중 graph regression, classification task에서 사용되는 TUDataset 중, ENZYMES 데이터 셋을 불러와보자
- ENZYMES 데이터 셋은 600개의 그래프와 총 6개의 레이블, 3개의 node feature로 구성되어 있는 데이터 셋이다.
from torch_geometric.datasets import TUDataset
import matplotlib.pyplot as plt
from collections import Counter
dataset = TUDataset(root = '/tmp/ENZYMES', name = 'ENZYMES')
## 출력 결과
Downloading https://www.chrsmrrs.com/graphkerneldatasets/ENZYMES.zip
Extracting /tmp/ENZYMES/ENZYMES/ENZYMES.zip
Processing...
Done!print(f'length of enzymes dataset : {len(dataset)}')
print(f'number of enzymes classes : {dataset.num_classes}')
print(f'number of node features : {dataset.num_node_features}')
## 출력 결과
length of enzymes dataset : 600
number of enzymes classes : 6
number of node features : 3def label_distribution(lb_cnt):
plt.bar(lb_cnt.keys(), lb_cnt.values())
plt.ylim(0, 150)
for grade, num in lb_cnt.items():
plt.text(
grade, num, num,
fontsize = 11, color = 'black',
horizontalalignment = 'center',
verticalalignment = 'bottom'
)
plt.show()
## data.y는 텐서값으로 되어 있는데, item() 메소드를 이용하여 레이블 값만 가져온다.
labels = [data.y.item() for data in dataset]
label_cnt = Counter(labels)
label_distribution(label_cnt)
## 출력 결과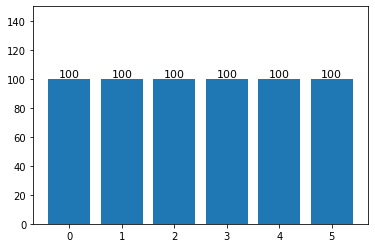
- Enzymes 데이터 셋중 아무 그래프나 하나 선택해서 시각화해보고, 어떤 특징을 갖는지 확인해 보자
- torch_geometric 형식의 그래프를 matplotlib에서 시각화하기 위해서는 networkx 형태로 변환해 그려줘야 한다.
from torch_geometric.utils import to_networkx
import networkx as nx
## 그래프 시각화 해주는 함수
def visualize_graph(graph):
plt.figure(figsize = (7, 7))
plt.xticks([])
plt.yticks([])
G = to_networkx(graph, to_undirected = graph.is_undirected())
nx.draw_networkx(G, pos = nx.spring_layout(G, seed = 0), with_labels = False, cmap = 'Set2')
plt.show()
sample_graph = dataset[222]
## 역시 되게 심오하게 생겼다..
visualize_graph(sample_graph)
## 출력 결과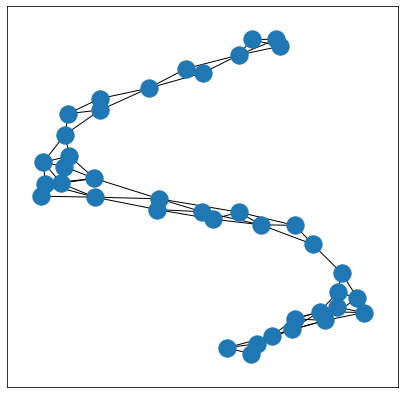
- 데이터를 확인해 본 결과, 158 / 2 = 79개(∵ undirected graph)의 edge와 40개의 node,
3개의 node feature가 있는 것을 확인하였다.
print(f'sample graph : {sample_graph}')
print(f'is sample graph undirected? : {sample_graph.is_undirected()}')
## 출력 결과
sample graph : Data(edge_index=[2, 158], x=[40, 3], y=[1])
is sample graph undirected? : True- 데이터 셋을 슬라이싱하여 데이터 셋을 학습용과 시험용으로 분할할 수 있다.
💬 개인적으로는 scikit-learn의 train_test_split을 활용하는게 제일 익숙하고 편하다..
import torch
test_size = int(len(dataset) * 0.3)
test_dataset = dataset[ : test_size]
train_dataset = dataset[test_size : ]
print(f'length of train dataset : {len(train_dataset)}')
print(f'length of test dataset : {len(test_dataset)}\n')
## 공식 홈페이지에서 소개하고 있는 데이터 셋을 셔플 하는 방법은 두 가지가 있다.
shuffled_dataset = dataset.shuffle()
sample_graph = shuffled_dataset[222]
visualize_graph(sample_graph)
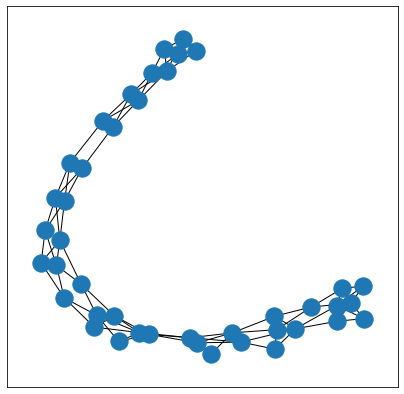
perm = torch.randperm(len(dataset))
shuffled_dataset2 = dataset[perm]
sample_graph = shuffled_dataset2[222]
visualize_graph(sample_graph)
## 출력 결과
length of train dataset : 420
length of test dataset : 180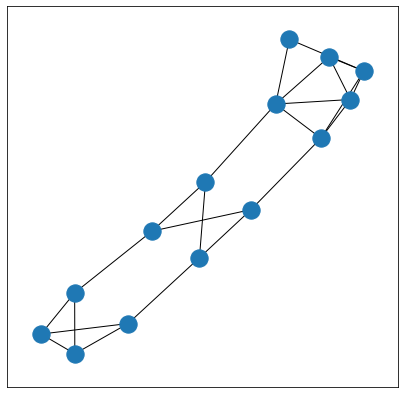
## 하지만 셔플해서 분할했다간... 균등한 데이터 분포라고 하더라도 특정 레이블만 학습용 데이터 셋에 많이 들어갈 수 있다...
shuffled_train = shuffled_dataset[test_size : ]
shuffled_test = shuffled_dataset[ : test_size]
train_lb_cnt = Counter([lb.y.item() for lb in shuffled_train])
test_lb_cnt = Counter([lb.y.item() for lb in shuffled_test])
label_distribution(train_lb_cnt)
label_distribution(test_lb_cnt)
## 출력 결과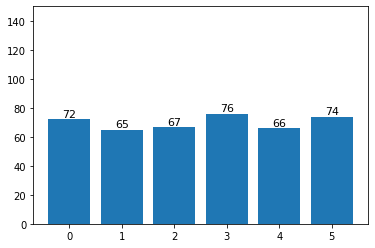
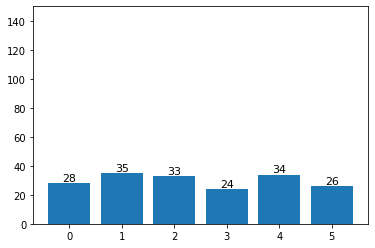
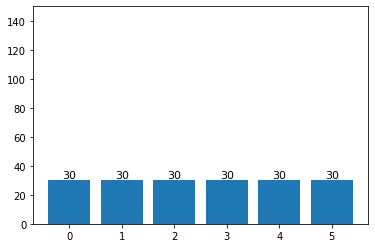
## 이번엔 scikit-learn에서 제공하는 방법으로 분할해보자.
from sklearn.model_selection import train_test_split
## scikit-learn의 train_test_split에서 stratify 옵션값으로 레이블들을 입력해주면, 레이블의 비율에 맞게 들어간다.
train_graphs, test_graphs = train_test_split(dataset, test_size = 0.3,
stratify = labels, shuffle = True)
train_lb_cnt = Counter([lb.y.item() for lb in train_graphs])
test_lb_cnt = Counter([lb.y.item() for lb in test_graphs])
label_distribution(train_lb_cnt)
label_distribution(test_lb_cnt)
## 출력 결과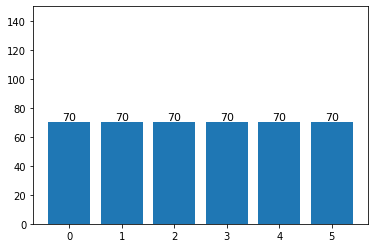
3-3. 미니 배치(Mini batch)
- PyG에서는 파이토치와 동일하게 DataLoader를 이용해 데이터 셋을 배치 단위로 나눌 수 있다.
from torch_geometric.data import DataLoader
loader = DataLoader(train_graphs, batch_size = 16, shuffle = True)
sample_batch = next(iter(loader))
print(f'sample batch : {sample_batch}')
print(f'number of sample batch graphs : {sample_batch.num_graphs} ')
## 출력 결과
sample batch : DataBatch(edge_index=[2, 1824], x=[547, 3], y=[16], batch=[547], ptr=[17])
number of sample batch graphs : 16
/usr/local/lib/python3.7/dist-packages/torch_geometric/deprecation.py:12: UserWarning: 'data.DataLoader' is deprecated, use 'loader.DataLoader' instead
warnings.warn(out)99. 참고자료
99-1. 도서
99-2. 논문, 학술지
99-3. 웹 사이트
- 아기여우의 자기계발 블로그 : Graph Neural Networks (GNN) / 그래프 뉴럴 네트워크 기초 개념 정리 | [블로그 링크]
- SuperMemi's Study : [CVPR2022 / GML4VC] 5. Pytorch Geometric 이란 무엇인가? | [블로그 링크]
- Doyeon's blog : GNN / Node2Vec and Latent Factor Model | [블로그 링크]
- Pytorch-geometric : INTRODUCTION BY EXAMPLE | [자료 링크]
- Heath.log : Node Embedding (정점 표현) | [블로그 링크]
99-4. 데이터셋 출처
전체코드
GitHub - EvoDmiK/TIL: Today I Learn
Today I Learn. Contribute to EvoDmiK/TIL development by creating an account on GitHub.
github.com
내용 추가 이력
부탁 말씀
개인적으로 공부하는 과정에서 오류가 있을 수 있으니, 오류가 있는 부분은 댓글로 정정 부탁드립니다.
728x90